Ressources
Le projet Populus
GÉNOME à l’École » propose aux enseignants et à leurs élèves de travailler sur le projet Populus, véritable formation à la recherche, consistant en l’observation de la diversité morphologique sur le terrain et l’analyse du polymorphisme nucléotidique de gènes d’intérêt chez le peuplier noir après séquençage de produits PCR (Polymerase chain Reaction).

Chaque lycée apporte sa contribution en travaillant sur une population de peuplier noir située à proximité de son implantation afin de construire collectivement une cartographie de la biodiversité du peuplier noir sur l’ensemble du territoire français.
Etude de la biodiversité à l’échelle moléculaire
Le séquençage de l’ADN de peuplier, réalisé grâce au matériel et aux partenaires de l’opération « GÉNOME à l’École », permet d’étudier les variations de l’ADN entre individus de peupliers noirs et entre les différentes espèces de peupliers présentes sur le territoire français (peuplier blanc, peuplier tremble)
Le cahier pédagogique
Les activités pédagogiques de biologie moléculaire peuvent se conduire dans des cadres institutionnels variés.
Afin de contribuer à l’échange et à la mutualisation des pratiques, les enseignants du réseau « GÉNOME à l’École » produisent des ressources pédagogiques et les proposent en accès libre. Les ressources ci-dessous ont été validées par le comité scientifique de l’opération « GÉNOME à l’École ».
Contexte avec la réforme du lycée général à la rentrée 2019-2020
Cette lecture des programmes proposée par Christian Tailliez (enseignant au lycée Bellevue du Mans, membre du comité scientifique de « GÉNOME à l’École ») permet de contextualiser les actions de l’opération dans les nouveaux programmes en vigueur en lycée général à partir de la rentrée 2019.
Nouveau contexte des actions GÉNOME avec la réforme du lycée général
Activité en classe de seconde générale
Proposition d’activité en classe de seconde générale dans le cadre du chapitre « Biodiversité, résultat et étape de l’évolution » par Christian Tailliez (lycée Bellevue du Mans).

partie 1 : concevoir une représentation graphique de la diversité des peupliers à partir de mesures biométriques simples effectuées sur diverses feuilles de peupliers
- Présentation
- Fichier « Biométrie» avec deux onglets : feuille vierge pour les élèves et feuille « corrigé » avec les feuilles choisies ici (versions PDF si lecture impossible : biométrie (correction) et biométrie (feuille vierge))
- Planches collection et cultivar
- Fiche méthode graphique Open Office
- Fichier choix pour bien choisir la feuille sur le rameau
partie 2 : élaborer une représentation de la diversité des arbres en considérant la diversité génétique observée sur une portion d’ADN
- Présentation
- Fichier « Div_peupliers.gpr » (Téléchargement sur demande à genome.ecole@observatoiredeparis.psl.eu)
- Fichier « séquence Genalys »
- Fichier « grille saisie SNP »
- Fichiers arbres « feuilles » et « distances »
Activité en classe de terminale, spécialité SVT
Proposition d’activité en classe de terminale, spécialité SVT, par Christian Tailliez (lycée Bellevue du Mans).
Questionner la notion d’espèce en s’appuyant sur les apports modernes du séquençage de l’ADN
- Présentation
- Documents : géographie_populus_allèles – recherche_enzyme_restriction – allèles
- Activité élèves
- Document : génotypes_populations
- Organisation matérielle
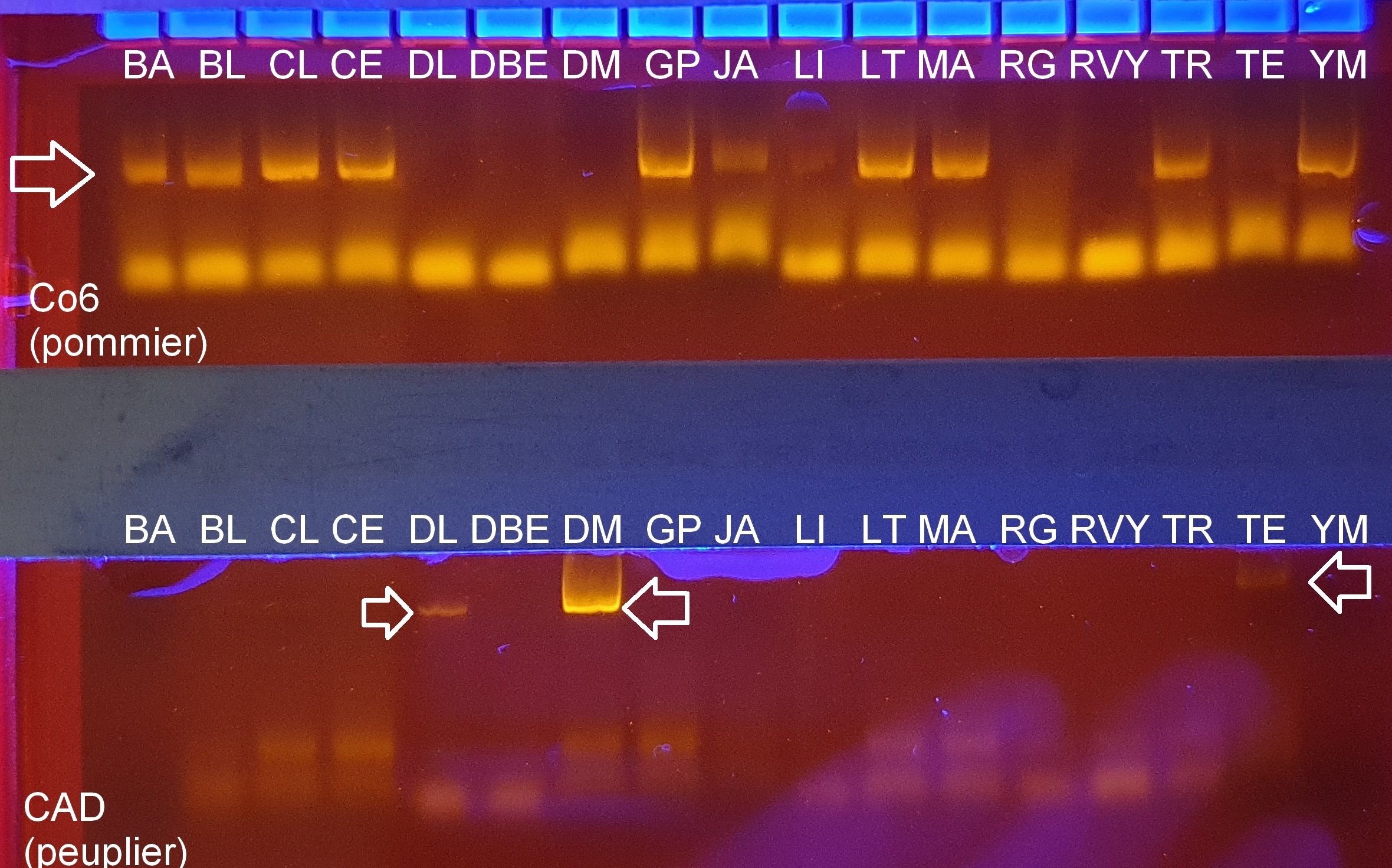
- Documents : résultats_attendus – résultats_électrophorèse
NOUVELLES MISES À JOUR – 26/06/2024
Tableurs collaboratifs du projet Populus
Proposition de tableurs collaboratifs initiés par Christian Tailliez (lycée Bellevue du Mans)


Depuis des années, qu’ils sont impliqués dans le projet Populus, de nombreux établissements ont accumulé des centaines de séquences et donc de génotypes. Les SNP responsables de la diversité présente dans les gènes ciblés dans le projet sont maintenant connus.
Comment valoriser tout le travail accompli, comment lui donner du sens ? En mutualisant les résultats obtenus par l’ensemble des lycées !
Ce qui est proposé ici est de rassembler, pour chaque gène, l’ensemble des résultats des lycées dans des tableurs collaboratifs, consultables en ligne ci-dessous pour tous les visiteurs de ce site.
Les données rassemblées dans ces tableurs permettent, avec les élèves, de comparer les populations des différentes régions françaises, de différentes latitudes, de tester Hardy-Weinberg, de formuler des hypothèses, d’évaluer les introgressions de deltoïdes ou d’italica etc…
Il ne s’agit pas de saisir obligatoirement les résultats pour tous les SNP, c’est pourquoi sont identifiés dans les tableurs cinq SNP prioritaires qui permettent de travailler :
- Tableur collaboratif Génotypes CRY2 France
- Tableur collaboratif Génotypes PhyA France
- Tableur collaboratif Génotypes PhyB1 France Mise à jour 26/06/2024
- Tableur collaboratif Génotypes PhyB2 France Mise à jour 26/06/2024
- Tableur collaboratif Génotypes CAD France Mise à jour 26/06/2024
Christian Tailliez propose le bilan d’activité qui reprend l’ensemble des données concernant CAD collectées dans le tableur collaboratif qu’il a mises mises en forme et analysées pour une part tout en proposant des problématiques. Ce bilan a vocation à (re)motiver un certain nombre de collègues dont ceux que le traitement des séquences a pu rebuter en les soulageant de cette tâche en leur transmettant des copies d’écran puisque sont maintenant connues les SNP intéressantes.
Ci-jointe également la fiche-carte (.doc et PDF) de ce travail accompli sur CAD depuis 10 ans.

Vous trouverez ci-dessous la carte des zones de prélèvement des peupliers. L’idée a été de cerner les bassins versants en distinguant la partie haute et la partie basse des fleuves afin de faire ressortir d’éventuelles hétérogénéités (amont vs aval, plaine vs montagne…)
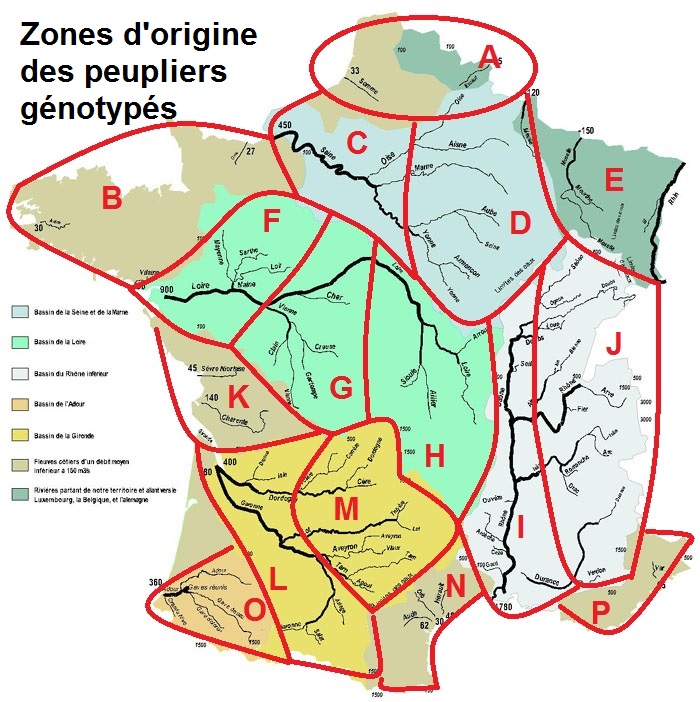
Les enseignants souhaitant apporter leur contribution à ce travail collaboratif peuvent télécharger les tableurs collaboratifs ci-dessus, les compléter puis les transmettre via l’adresse de contact ci-dessous. Toute contribution est la bienvenue !
Contact : genome.ecole@observatoiredeparis.psl.eu
Liste des établissements contributeurs :
Académie |
Nom de l’établissement |
Ville |
Contribution Génotypes |
|---|---|---|---|
| INRAE | CRY2 et PhyB1 | ||
| INRAE Guémené-Penfao | CRY2 | ||
| Bordeaux | Lycée Jay de Beaufort | Périgueux | PhyB2 |
| Créteil | Lycée Marcelin Berthelot | Saint-Maur-des-Fossés | PhyB2 |
| Grenoble | Lycée Marlioz | Aix-les-Bains | CAD, CRY2, PhyA, PhyB1 et PhyB2 |
| Limoges | Lycée Raoul Dautry | Limoges | PhyB2 |
| Nantes | Lycée Bellevue | Le Mans | CAD, CRY2, PhyA, PhyB1 et PhyB2 |
| Orléans-Tours | Lycée Jean Moulin | Saint-Amand-Montrond | CAD, PhyB1 et PhyB2 |
| Versailles | Lycée Jean-Pierre Vernant | Sèvres | CAD, PhyA, PhYB1 et PhyB2 |
 |
« Sciences à l’École » tient à saluer et remercier la précieuse collaboration de monsieur Philippe Poupart (pépinière expérimentale de l’Office National des Forêts à Guémené-Penfao) qui permet aux enseignants du réseau GÉNOME de disposer de boutures de peuplier noir. |
Les projets propres des établissements
Les équipes pédagogiques du réseau investissent leur savoir-faire expérimental acquis grâce au projet Populus dans un autre projet, de leur choix, dit projet propre, sur la base duquel elles ont été sélectionnées. Les thèmes, multiples et variés, ont souvent un lien avec le territoire d’implantation des établissements.
ENQUÊTE DE POLICE SCIENTIFIQUE AU LYCÉE BELLEVUE DU MANS
Par Christian Tailliez, professeur de SVT et membre du comité scientifique de l’opération « GÉNOME à l’École » :
« Qu’il s’agisse du projet Populus ou du projet propre notre démarche avec les élèves (en seconde essentiellement) est parfois ardue et une approche un peu ludique peut aider à entraîner tous les élèves dans l’aventure de l’ADN. Je vous propose une programmation de séances associant une classe de seconde générale et un groupe de première Spécialité SVT autour d’une enquête de Police Scientifique. Le travail mené n’exclut absolument pas le travail d’obtention de séquences nouvelles pour l’enrichissement des banques de séquences des projets… »
Résumé de la démarche
Chaque élève de la classe de seconde va être un technicien-enquêteur chargé d’extraire l’ADN de l’indice puis d’exploiter le résultat de la PCR (obtenu par un « collègue » de première…) pour enfin rédiger un rapport de conclusions. Un enjeu important de cette démarche est de faire toucher du doigt par chaque élève la réalité que l’ADN est détenteur d’information. Pour cela il est prévu que l’indice soit d’une espèce ou d’une autre selon l’élève-enquêteur, qu’une anonymisation des tubes après extraction soit réalisée et que la validité du résultat puisse être constatée a posteriori (voir ci-après). Pour le rapport laisser les élèves libres sur la forme et le fond et valoriser lors de la restitution des copies les bonnes initiatives !
Documents à télécharger en un clic !
- lenquete_1.pdf (distribué lors de la première séance)
- lenquete_2.pdf (distribué lors de la première séance)
- comprendre_laphoto.pdf (distribué lors des résultats PCR)
- électrophorèse 1SVT3.jpg
- PCR_enquete.jpg
- Planification_V2.pdf
Pour vérifier la validité du résultat a posteriori, deux possibilités :
- mettre deux feuilles (1 de pommier et 1 de peuplier) dans l’enveloppe initiale et l’élève choisit celle qui sera son indice. L’autre est laissée dans l’enveloppe qu’on scelle avec le code-barre et qu’on rouvre à la fin pour constater la cohérence du résultat PCR ;
- deux élèves se partagent un indice imposé (feuille de pommier ou feuille de peuplier) donc à la fin leur résultat PCR doit être le même…

SEXAGE GÉNÉTIQUE DU LYNX À PARTIR D’ÉCHANTILLONS FÉCAUX AU LYCÉE XAVIER MARMIER À PONTARLIER
Partenaires : Eve Afonso-Douablin, Maître de conférences universitaire ; Laboratoire Chrono-environnement UMR UFC/CNRS 6249 USC INRA ; Université de Franche-Comté.
Enseignants impliqués : Sumka/Aubry/Esseiva (SVT), lycée Xavier Marmier à Pontarlier.
Objectif
Le Jura (géologique) comprend la population de Lynx la plus abondante en France (estimée à 120 individus). Suite à la colonisation du massif Jurassien par une population de Lynx Suisses, une étude sur la diversité génétique de la population est initiée par l’UFR.
Divers marqueurs génétiques sont étudiés afin d’estimer cette diversité. L’analyse porte sur les « crottes » de Lynx (technique non invasive et non dérangeante).
L’étude génétique des crottes permet ainsi :
- de retrouver le régime alimentaire de l’animal ;
- d’avoir un aperçu de la diversité génétique ;
- de connaitre le Sex ratio.

Démarche
C’est dans le cadre de l’étude du sex ratio que nous travaillons sur des échantillons fournis par l’UFR. Des marqueurs spécifiques du chromosome Y et X ont été identifiés (Wei 2006 ) .


Lors de notre visite dans le laboratoire de notre partenaire, le kit d’extraction à utiliser nous a été indiqué et nous avons pu affiner le protocole d’extraction d’ADN et d’amplification des marqueurs sexuels.
Difficultés
Contrairement au Populus, l’extraction est plus délicate en raison des contaminations éventuelles. Nous avons ainsi affiné le protocole et la version 12 ( !!) semble désormais actée.
Résultats
Après un essai en effectif restreint (février 2022) , une extraction + amplification + contrôle par électrophorèse a été réalisée par l’équipe génome (12 élèves répartis de la seconde à la terminale). Les résultats envoyés à Mme Afonso ont été contrôlé et validés.

Résultat d’une partie de notre travail. 4 échantillons ont été testé en tout.
Perspectives
L’opération va donc être reconduite sur d’autres échantillons. Une ouverture sur un programme concernant le chat forestier va être engagé ensuite.

Une partie de l’équipe GÉNOME. Le mercredi après-midi, pas facile d’avoir tout le monde à la fois !
Crédits photos : N. ESSEIVA
ÉTUDE D’UNE RACE BOVINE RARE « LA BRETONNE PIE NOIR » AU LYCÉE MARCELIN BERTHELOT
« De 2016 à 2018, les élèves du lycée Marcelin Berthelot à Saint-Maur-des-Fossés (académie de Créteil), ont étudié le déterminisme génétique de phénotypes rares chez une race bovine à petits effectifs, la Bretonne Pie noir (BPN). Autrefois très répandue, et diverse, la BPN est passée près de l’extinction et la question de la diversité génétique résiduelle dans cette race bovine se pose. Nous avons été accueillis par Alain Divo, qui élève des BPN (parmi d’autres races bovines et caprines à petits effectifs) à Fontenay-lès-Briis (Essonne) : dans sa ferme, les élèves ont prélevé les poils de plusieurs individus aux phénotypes remarquables, afin d’en extraire l’ADN. Nous avons également bénéficié de l’expertise d’Aurélien Capitan, chercheur à l’INRA. Le projet s’est décliné en deux volets :
Volet « culard »
Des élèves de première S ont travaillé sur une vache musculeuse au phénotype « culard », jamais encore décrit dans cette race (2 autres culards l’ont été depuis). Le phénotype culard est causé par le défaut ou l’absence d’une protéine nommée myostatine, lorsque les deux copies du gène correspondant (MSTN) sont mutées. Nous voulions savoir s’il s’agissait d’une des 9 mutations déjà connues dans MSTN, et qui aurait été introduite dans la race BPN à la faveur d’un croisement, ou bien d’une mutation nouvellement apparue.
Noé, taureau BPN culard dans l’élevage d’Eric Tocqueville
À la découverte des Bretonne pie noir (pas forcément noires !) dans l’élevage d’Alain Divo
Encadrés par les enseignants du LMB Aurélien Terracol et Caroline Nedellec, les élèves ont séquencé l’ensemble de la séquence codante du gène MSTN (répartie sur 3 exons). Ils ont découvert dans l’exon 3 une mutation non-sens jamais encore décrite. Cette nouvelle mutation a pour conséquence la production d’une protéine myostatine tronquée, sans doute inactive. Par analyse du pédigrée de l’animal culard, Aurélien Capitan a pu remonter à l’origine de la mutation : un taureau qui est l’arrière-grand-père de notre vache cularde du côté paternel, et son quadrisaïeul du côté maternel. Comme l’on connait la contribution de ce taureau à la race BPN, on peut estimer la fréquence de l’allèle muté dans la population, donc celle des hétérozygotes (chez qui la mutation a un effet positif sur la carcasse, négatif sur la note d’engraissement), ainsi que la probabilité d’apparition d’un nouveau culard (mutation à l’état homozygote).
La découverte de cette nouvelle mutation intéresse les éleveurs de BPN (regroupés dans l’Union Bretonne Pie Noir). Notre recherche a donc un débouché pratique. Nos élèves ont pu s’initier au monde de la recherche, mais aussi découvrir les problématiques de l’élevage.
Volet « robe »
Autrefois, les vaches BPN n’avaient pas toutes une robe noire. Certaines étaient « rouges », d’autres gris-bleu (« glazik ») ou chamoisées. Lors de la résurrection de la BPN dans les années 1970, les éleveurs ont mis à contribution un petit nombre de reproducteurs conformes au standard de race (robe pie noir) établi en 1884. Toutefois des individus de robe rouge ou glazik apparaissent encore et sont présents dans l’élevage d’Alain Divo.
Un veau « glazik » et sa mère chamoisée dans l’élevage d’Alain Divo
Analyse des séquences avec les enseignants et les élèves de BCPST et de première S le 28 juin 2018
Encadrés par les enseignants Vincent Lun et Yann Esnault, des élèves de BCPST du lycée Berthelot ont d’abord exploré le locus Extension (gène MC1R) qui est le premier déterminant de la couleur de robe (il détermine la balance entre le pigment noir – eumélanine – et le pigment brun – phéomélanine). Nous avons confirmé que la couleur noire est due à l’allèle dominant ED comme chez les autres races noires bovines ; mais nous avons également établi la présence de l’allèle E+ qui, à l’état homozygote, donne une robe « rouge ».
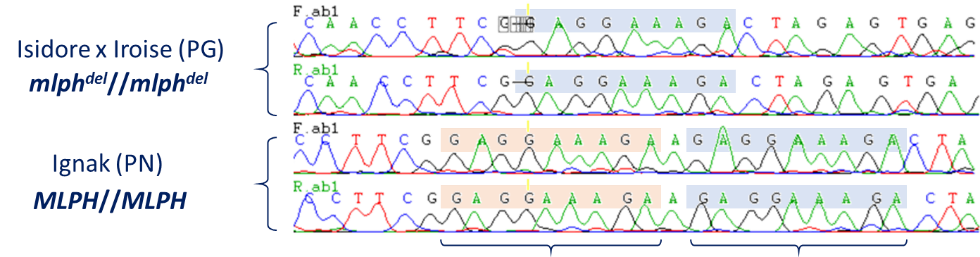
Nous avons ensuite recherché la cause de la robe « glazik » par une approche de type « gènes candidats ». Nous avons exploré quatre gènes déjà connus pour des mutations responsables de couleurs rares, par l’utilisation d’amorces ad hoc. Nous avons retrouvé, dans le gène MLPH, une délétion déjà décrite chez la race Blanc bleu belge et responsable dans cette race d’une robe « diluée » à l’état homozygote. Le gène MLPH code la mélanophiline, une protéine responsable de la répartition dans l’épiderme et le poil des pigments (mélanines) produites par les mélanocytes. Aurélien Capitan à l’INRA a confirmé le lien causal entre la délétion et la couleur de robe bleu-gris dans la BPN par l’analyse de marqueurs génétiques. Là aussi, ce résultat donne un outil aux éleveurs BPN pour connaître le potentiel de leur cheptel en matière de couleur de robe.
La délétion responsable de la robe « glazik » mise en évidence dans le premier exon codant du gène MLPH au niveau d’une duplication
Un article scientifique présentant ces résultats est en cours d’écriture. Une présentation plus détaillée des travaux des élèves du lycée Berthelot est accessible ci-dessous. Depuis 2018, l’atelier Génome se poursuit au lycée Berthelot : après avoir travaillé sur les variétés anciennes de pommiers en 2019 et 2020, nous travaillons désormais sur la diversité des cafés (genre Coffea) et les gènes de biosynthèse de la caféine. »
Yann Esnault, enseignant en CPGE, prépa biologie, chimie, physique et sciences de la Terre,
lycée Marcelin Berthelot
SAUVEGARDE DE LA BIODIVERSITÉ DOMESTIQUE RÉGIONALE AU LYCÉE JEAN MOULIN

Un projet très ambitieux utilisant le matériel de l’opération « GÉNOME à l’École » a été mis en place dès l’année 2011 par Véronique Ranty et Perrine Girard, professeures de SVT au lycée Jean Moulin à Saint-Amand-Montrond (académie d’Orléans-Tours). Ce projet s’articule sur la collaboration avec l’URGC : l’Union pour les Ressources Génétiques du Centre-Val de Loire.
Le partenariat avec Pierre Picot, viticulteur local, a été l’élément déclencheur qui a permis au lycée d’intégrer le réseau d’établissements de l’opération « GÉNOME à l’École » en 2011. Ce producteur envisage alors de redonner vie à un cépage ancien et oublié : le gouget. Sa démarche commerciale est basée sur la mise en valeur des produits du terroir, en s’appuyant sur l’histoire et la culture qui leur sont associées : le vin issu de ce cépage était produit dans la région et servi à la table de Louis XIV.
Au secours d’un cépage oublié : le gouget !
Différents groupes d’élèves de première et terminale ont travaillé pendant trois ans pour rendre possible la production commerciale de vin issu de gouget : recherche de ceps de gouget supposé dans des vignes de particuliers, identification ampélographique, identification génétique, essais de micro vinification et identification des démarches à suivre pour sa réhabilitation commerciale.
Plantation de ceps à Vesdun au printemps 2017 (lien vidéo journal France 3)
A partir de 2017, le projet gagne encore en ampleur. D’autres établissements scolaires s’associent à la démarche en s’appuyant sur la réhabilitation de légumes anciens, autour de plusieurs opérations pilotes : challenge culinaire, jardins pédagogiques, sciences participatives (PL@NTNET)
Entre 2019 et 2021, une convention de partenariat signée entre l’académie d’Orléans-Tours et l’URGC dynamise le projet avec plusieurs objectifs : sensibiliser les élèves à la sauvegarde des races et variétés domestiques anciennes de la région Centre-Val de Loire et faire contribuer les élèves à la promotion et à la consommation des variétés anciennes sauvegardées.
BARCODING MOLÉCULAIRE AU LYCÉE ÉLISA LEMONNIER

Un projet utilisant le matériel de l’opération « GÉNOME à l’École » a été mis en place à la rentrée scolaire 2018 par Farida El Mallouli, professeure de Biotechnologies Biochimie génie biologique au lycée Elisa Lemonnier à Paris, en partenariat avec l’Aquarium Tropical du Palais de la Porte Dorée (Gabriel Picot, Chargé de développement culturel et pédagogique) et le Muséum national d’Histoire naturelle (Agnès Dettaï, Maître de Conférences)
Celui-ci consiste en l’étude du génome de spécimens de poissons de l’Aquarium, avec une nouvelle technique : le barcoding moléculaire.
Lors de la Fête de la science en octobre 2019, un très jeune public, venu à l’Aquarium Tropical, a pu découvrir cette technique :

Lors de l’année de mise en place du projet (2018-2019), les élèves du lycée Elisa Lemonnier ont testé différentes espèces de poissons issus des bassins de l’Aquarium Tropical. La galerie ci-dessous présente les photos de quelques-uns des individus qui ont été étudiés par les élèves.
À la rentrée 2019, le projet s’est affiné : les lycéens de Terminale STL ont été amenés à se focaliser sur l’étude des populations de raies des bassins de l’Aquarium Tropical. Au programme des activités : prélèvement d’échantillons, extraction d’ADN, amplification en chaîne par polymérase (PCR), puis électrophorèse sur gel d’agarose. Le séquençage est réalisé par le Genoscope d’Evry, partenaire de « GÉNOME à l’École ».
L’objectif est de permettre l’identification de l’espèce, la détermination de la provenance géographique des individus et l’établissement de la filiation entre individus. Durant deux années scolaires, ce sont des dizaines d’individus d’espèces différentes qui ont pu être étudiés :
Extraction de l’ADN, amplification en chaîne par polymérase puis électrophorèse sur gel d’agarose
En 2020-2021, le projet se poursuit. Les ressources pédagogiques ci-dessous donnent une idée de la démarche envisagée et de l’ambition des enseignants porteurs du projet au lycée Elisa Lemonnier, au travers d’une sélection d’activités documentaires et expérimentales et de documents d’accompagnement :